芸苔属基因组染色体级比较分析研究
芸苔属(Brassica)物种在农业生产和进化研究中具有重要地位,其基因组受到多倍化事件和广泛结构变异的影响。在发表于《植物分子生物学》(Plant Molecular Biology)的最新研究中,我们发布了黑芥(Brassica nigra)的染色体级基因组组装序列,并对芸苔属染色体进行了比较分析,揭示了进化分化与结构变异的特征。
主要发现
-
高质量黑芥基因组组装: 利用 Hi-C 测序技术,我们生成了 484 Mb 的组装序列,其中 393 Mb 成功锚定到 8 条染色体上,显著提升了以往参考基因组的分辨率和基因注释准确性。
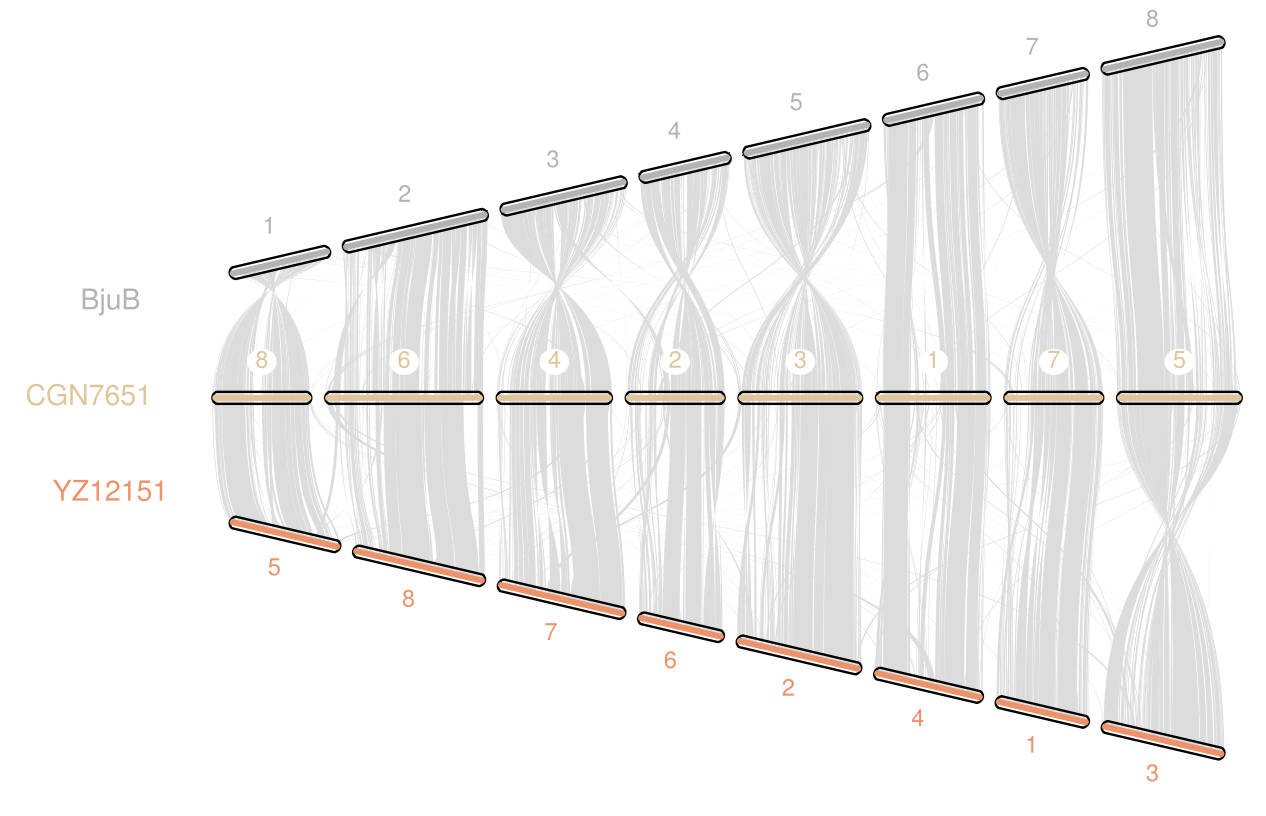
- 芥菜类作物 B 基因组的比较分析:
- 将组装的黑芥基因组与芥菜(B. juncea)中的 B 亚基因组进行对比,揭示了多倍化后显著的染色体变异。
- 发生剧烈变异的区域表现出较低的基因密度和较高的转座子(TE)含量,强化了基因组结构在塑造物种分化中的作用。
- 芸苔属染色体分化的进化洞察:
- 对芸苔属 A、B、C 基因组的染色体比较制图表明,A 基因组与共同祖先更为接近,而 C 基因组在分化后经历了更多的结构变异。
- 鉴定出三种芸苔属二倍体之间的同源区域,深化了我们对芸苔属物种染色体进化和基因组可塑性的理解。
研究感悟
我刚加入华大(BGI)时就认识了王文亮,多年来他已成长为一名专注的生物信息学专家。这项研究是他博士学位论文的核心内容,是与我们武汉的合作伙伴共同完成的。
该项目启动之初,我们愿景是利用新开发的测序平台开展大规模基因组研究,并选择了黑芥这一重要但此前代表性不足的物种进行群体基因组学研究。面对其 10 Gb 基因组规模带来的计算挑战,重测序与组装工作需要借助 Hi-C 挂载等先进的支架构建技术。通过克服这些技术障碍,我们成功完善了基因组组装和进化分析,为芸苔属基因组学及育种应用提供了宝贵的资源。
研究全文可通过 Plant Molecular Biology 在线访问。