抹香鲸染色体级基因组组装:洞察海洋哺乳动物进化
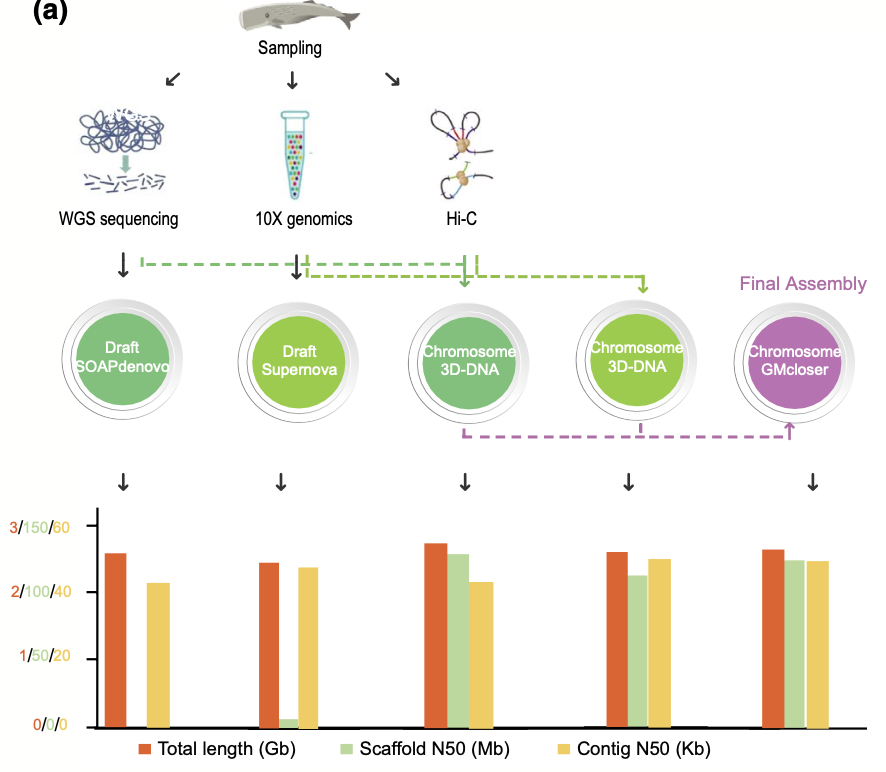
海洋哺乳动物是独特的进化模型,展示了通过生理和遗传修饰对水生环境的适应。其中,抹香鲸(Physeter macrocephalus)作为潜水最深的海洋哺乳动物之一,因其极端的生理能力一直吸引着科学家的关注。在发表于《分子生态学资源》(Molecular Ecology Resources)的最新研究中,我们利用自主研发的高通量测序平台 BGISEQ-500 产生的数据,发布了首个抹香鲸染色体级基因组组装序列,并探讨了其染色体进化、重复序列含量、基因家族动态以及对海洋环境的遗传适应。
主要发现
- 高质量染色体级基因组组装:
- 我们结合使用 BGISEQ-500 测序、10X Genomics 和 Hi-C 挂载技术成功组装了抹香鲸基因组,实现了 121.90 Mb 的 Scaffold N50,并将 94.34% 的序列锚定到 21 条染色体上。
- 比较分析显示了极高的基因组连续性,通过将 99.29% 的墨西哥湾抹香鲸组装序列比对到我们的参考基因组,更有效地解决了缺漏(Gaps)和重复区域问题。
- 染色体进化与结构重排:
- 通过重建祖先染色体,我们追踪了抹香鲸与其陆地近缘物种(如牛)之间的主要染色体间重排,检测到了鲸目动物进化中独特的染色体断裂(Fission)和融合(Fusion)事件。
- 鉴定出与缺氧适应、氧气保存和深潜能力相关的基因扩张,揭示了抹香鲸生理特性的遗传基础。
- 海洋适应与正向选择:
- 功能富集分析揭示了涉及氧气运输、抗应激和代谢适应的正向选择基因(PSGs),突显了支持长时深潜和极端深度耐受性的遗传路径。
- 研究证实了免疫相关通路中的基因家族收缩,表明其具有适应海洋环境的独特病原体抗性机制。
研究感悟
在自主研发的高通量测序平台发布后,我们旨在通过对代表性物种进行基因组测序和组装来展示其性能,这与我们之前的大熊猫基因组项目类似。抹香鲸基因组项目正是这一努力的体现,它利用了 BGISEQ-500 数据进行组装。鉴于抹香鲸是哺乳动物,基因组组装相对成功,提供了高分辨率的染色体级洞察。然而,与依赖长读长测序技术(Long-read sequencing)的组装结果相比,仍存在一定的局限性,这仍是未来优化基因组组装策略需要面对的挑战。
研究全文可通过 Molecular Ecology Resources 在线访问。