利用长片段读取测序实现人类三元组的高精度单倍型组装
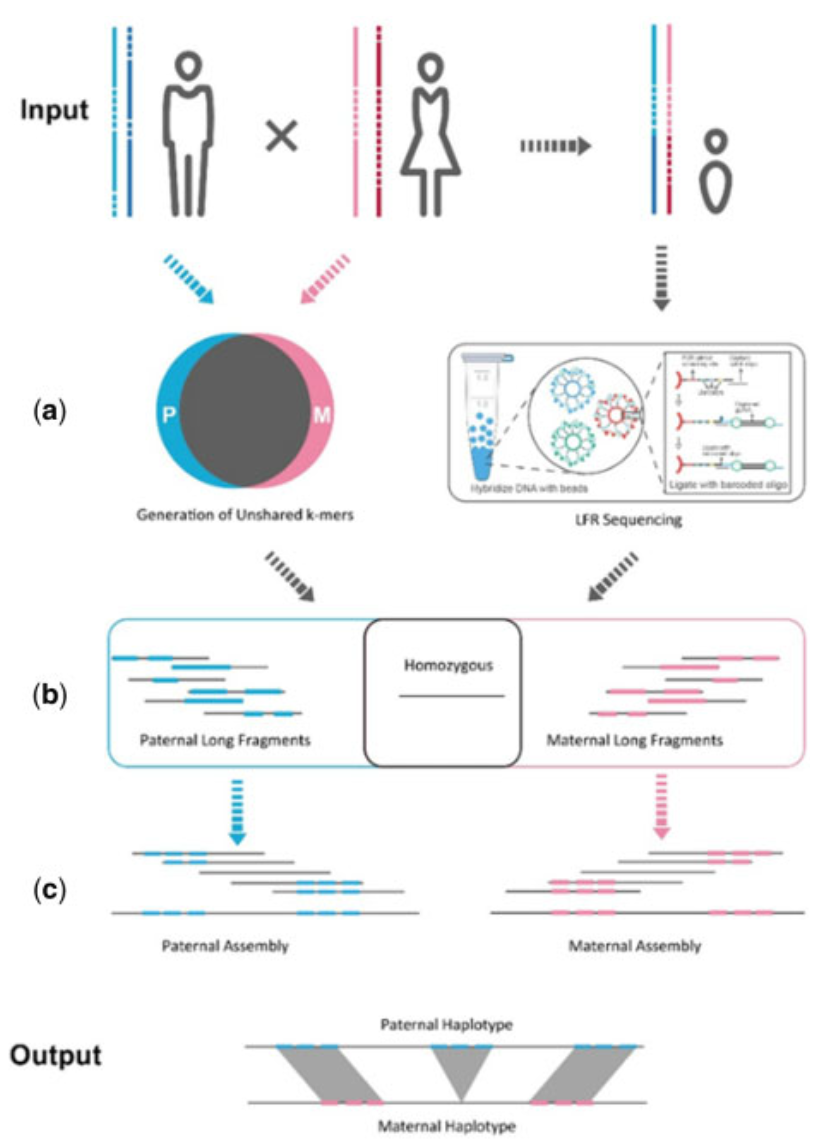
了解遗传变异如何在个体基因组内遗传和分布,是推进精准医学和进化研究的关键。在发表于《Bioinformatics》的最新研究中,我们提出了 HAST(基于三元组分箱策略的合成长读长单倍型组装,Haplotype-resolved Assembly for Synthetic long reads using a Trio-binning strategy)。该方法利用长片段读取(LFR)测序技术,显著提升了人类三元组(Trio)样本单倍型组装的准确性。
主要发现
-
改进的三元组单倍型组装: 我们开发了 HAST,这是一种能够高效解析三元组测序数据的全新计算流程。通过整合双亲的基因组信息,HAST 成功地对长基因组序列进行了相位分析(Phasing),将单倍型精准度提高至约 99.7%,召回率提升至约 95.9%。
-
利用 LFR 实现高质量基因组组装: 基于 LFR 测序技术(最初由 Complete Genomics 推出),我们将其应用于人类三元组。LFR 测序与 Hi-C 数据的结合实现了染色体级别的基因组组装,从而更精确地重建了父系和母系单倍型。
-
结构变异与单碱基准确度: 与 TrioCanu 等其他三元组分箱方法相比,HAST 展示了卓越的单碱基准确度(最高可达 Q65),同时有效地保留了结构变异(SVs)。该方法在检测复杂基因组重排方面表现尤为出色,而传统的基于比对的单倍型分析往往会遗漏这些变异。
研究感悟
这项研究标志着我们在利用 LFR 测序技术处理人类三元组数据集并实现单倍型组装方面迈出了重要一步。Complete Genomics 是最早引入 LFR 技术的公司之一,此前已在《Nature》等期刊上展示了该技术在单倍型相位分析中的作用。LFR 测序的核心理念类似于 BAC 库测序——能够跨越长基因组片段有效地进行序列重建,是单倍型组装的理想方法。
在本研究中,我们整合了多种基因组数据集以精炼单倍型重建过程,最终生成了可作为测序标准(Sequencing standards)的单倍型解析组装结果,用于评估其他基因组组装和分析工具。虽然 LFR 测序面临读长限制等挑战,但我们的研究证明了其在生成高精度单倍型组装方面的有效性。这项工作的顺利完成离不开团队成员及合作伙伴的辛勤付出。
研究全文可通过 Bioinformatics 在线访问。