SLR-superscaffolder:一种针对合成长读长的全新基因组挂载工具
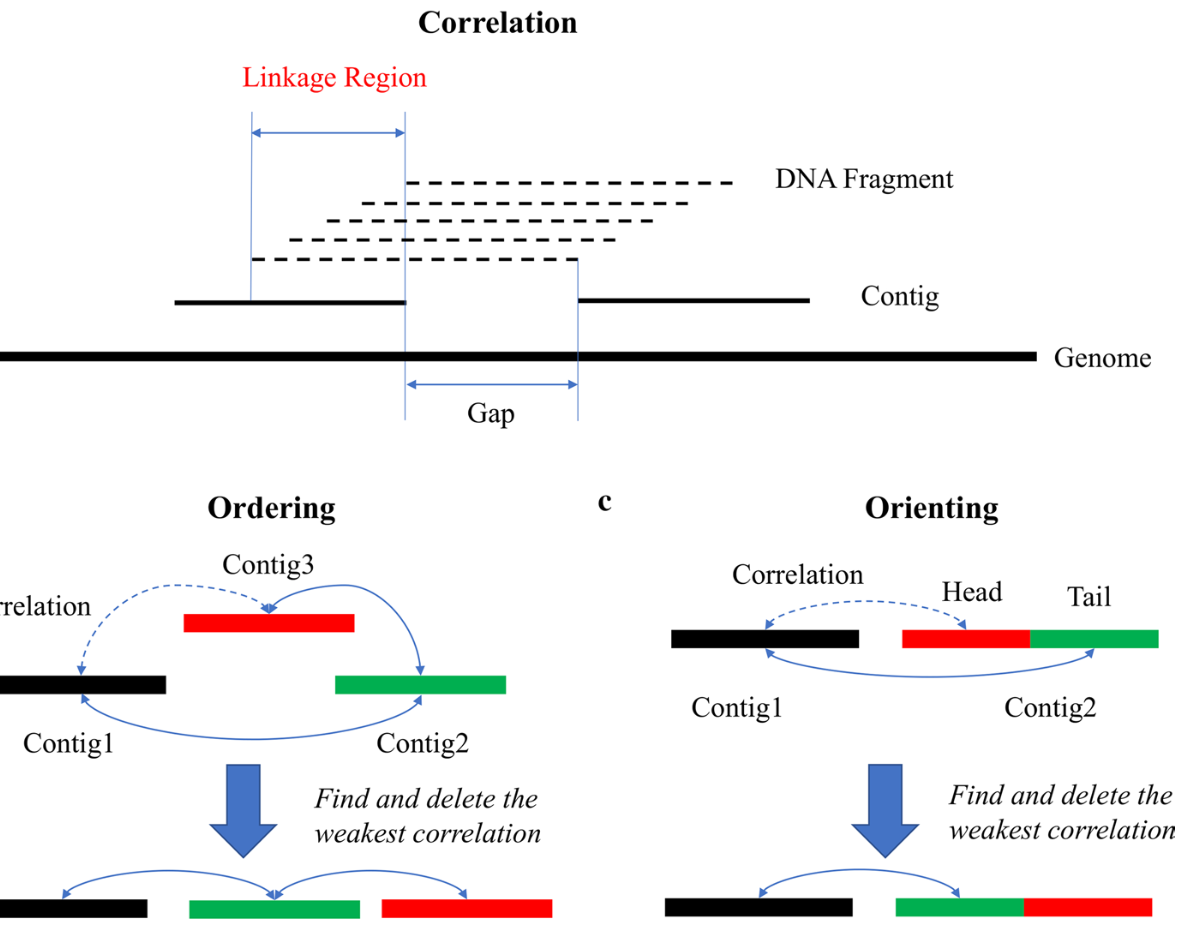
基因组组装是基因组学研究的基石,它能够将原始测序数据重建成完整的遗传序列。随着测序技术的进步,合成长读长(Synthetic Long Reads, SLR)已成为提高组装质量的一种高效策略。在发表于《BMC Bioinformatics》的最新研究中,我们推出了 SLR-superscaffolder。这是一款功能强大的独立挂载(Scaffolding)工具,旨在利用共条形码(Co-barcoding)和双端测序数据显著提升组装的连续性。
主要发现
-
针对合成长读长的独立挂载工具: SLR-superscaffolder 整合了共条形码信息和双端测序信息,将初步组装的 Contigs(重叠群)连接成 Scaffolds(支架),从而大幅提升基因组组装的连续性。
-
创新的“自顶向下”算法: 与以往的挂载工具不同,我们的算法首先基于 Jaccard 相似度(Jaccard Similarity)构建全局挂载图(Global scaffold graph),在进行局部优化前先确定 Contigs 的顺序和方向。这种层次化方法有效减少了基因组组装中常见的错误和误组装。
-
优于现有挂载工具的稳健性能: 我们将 SLR-superscaffolder 与 fragScaff、Architect 和 ARKS 等主流挂载工具进行了基准测试。结果显示,我们的工具在取得更长的 Scaffold NG50 值的同时,误组装数量更少。
-
在人类基因组组装中的应用: 该工具被应用于人类基因组测序数据集(HG001),将 Scaffold NG50 提升了 1349 倍,证明了其在大规模基因组项目中的实用性。
研究感悟
基因组组装算法是生物信息学研究的核心。在华大基因(BGI),特别是 2014 年之后,我们的重点转向了开发基于自主测序平台的技术。然而在那段时期,长读长测序(Long-read sequencing)因其卓越的连续性和准确性而在组装领域备受青睐。从实验和计算的角度来看,长读长测序简化了组装过程,并能产生更高质量的结果。
但当时我们的平台大多是为短读长测序优化的,这使得组装工作更具挑战性。幸运的是,我们开发了单管长片段读取(stLFR)测序技术,其原理类似于在短读长平台上实现 BAC 文库测序。这种方法为通过挂载技术改善组装质量提供了有效途径。在本研究中,我们利用 stLFR 测序数据进行挂载,成功增强了基因组组装的质量和连续性。这项研究是将短读长测序与先进组装策略相结合的重要一步。
研究全文可通过 BMC Bioinformatics 在线访问。